丨全基因组进化树及遗传多样性分析
通过对26个样本进行三代+二代测序,该研究成功组装了32条完整的HAdVs基因组序列,被归类为HAdV-B、C和E三类。进化分析揭示了HAdV-B类包含B3、B7和B55三个基因型,而HAdV-C类则包含C1、C2和C5三个基因型。值得注意的是,S9样本中存在两条35 Kb左右的长contig,且被划分为不同的系统发育分支,表明它们的进化轨迹存在差异,并且提示S9样本可能存在混合共感染的情况。在遗传多样性方面,该研究发现HAdV-C在六邻体基因(hexon)、纤维基因(fiber)和E3基因区域中展现出显著的高多样性。在基因组的其他区域,HAdV-C的遗传多样性低于HAdV-B。
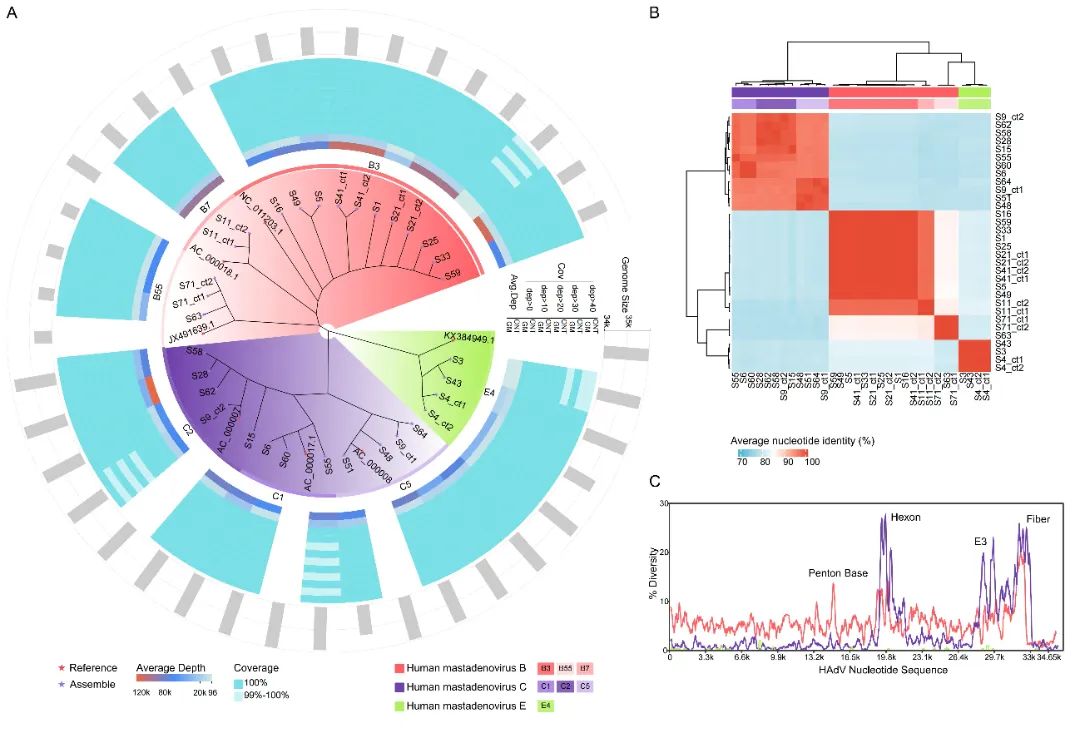
图1 HAdVs全基因组进化树及多样性分析
丨S9样本混合感染的验证
通过计算机模拟限制性内切酶分析,该研究发现S9样本的两条contig与已知的HAdV-C5和HAdV-C2参考基因组存在明显的遗传相似性。随后的序列比对分析显示,全基因组同源性分别高达98.68%和99.69%,并且在关键病毒核衣壳基因(Hexon, Penton, Fiber)序列中存在很高的相似性。以上结果证实了S9样本中腺病毒混合感染的复杂性,还展示了混合组装的优势,对于病毒菌株的精确基因分型和揭示病毒感染的复杂性至关重要。

图2 S9样本混合感染研究
丨腺病毒基因的一致性及多样性分析
HAdV-B、C和E中,Hexon、Fiber、E3基因的进化树分析结果与全基因组高度一致,而在Penton、E1A、E1B、DNA pol和E4基因区域,HAdV-C显示出较低的遗传多样性,未形成清晰的分支。核苷酸一致性分析进一步确认了HAdV-C在E1A、E1B、DNA pol、Penton和E4基因区域的高度遗传保守性,而Hexon、Fiber和E3基因区域则表现出较低的核苷酸同源性和较高的多样性。同义/非同义突变分析显示,HAdV-B和HAdV-C的大部分基因Ka/Ks比小于1,表明它们主要处于纯化选择下。然而,HAdV-C E4基因的Ka/Ks比值大于1,暗示存在正选择作用,增强病毒对宿主的适应性。这种增强可能是通过调节宿主细胞功能、促进病毒DNA复制及RNA加工过程来实现的。

图3 HAdVs基因多样性、一致性及突变情况分析
丨重组分析
重组是HAdV进化的主要驱动力之一,可能影响病毒的宿主适应性和传播能力。在本次组装得到的基因组中,HAdV-C的重组网络比HAdV-B更为复杂,并且识别出了更多的重组事件。重组断点遍布整个基因组,特别是在HAdV-C中,Hexon和Fiber基因区域存在两个明显的重组热点。此外,S9样本的重组分析显示,S9-contig1可能是一个涉及C5参考菌株和S9-contig2的重组序列。

图4 HAdVs重组分析
丨共线性和GC含量分析
共线性分析揭示了HAdV-B和HAdV-C同种内菌株序列的高度相似性,这一发现为理解腺病毒的遗传稳定性提供了重要线索。GC分析表明,基因组中容易发生同源重组的区域,GC含量明显较低,特别是在Hexon、E3、Fiber和E4基因区域。这些区域不仅核苷酸多样性高,而且还是重组的热点区域。
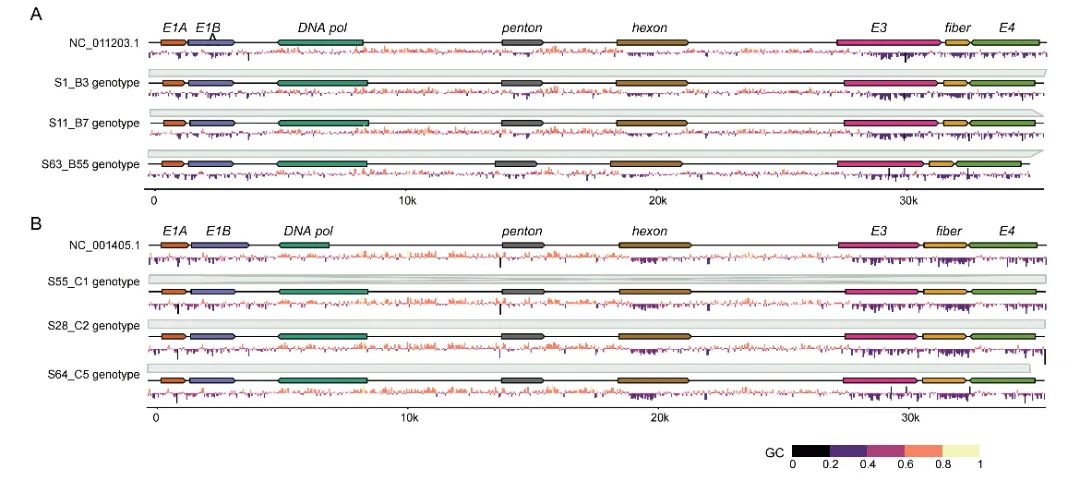
图5 HAdVs共线性及GC含量分析